塩基編集(Base Editing)の仕組みをわかりやすく解説【CRISPR-Cas9を発展させた切らないゲノム編集】
CRISPER-Cas9は、DNAの二本鎖を切断することでゲノムを編集する技術です。
狙った箇所でDNAを切断し、狙った配列を取り除くことができます。
詳しくはこちらの記事に書いていますが、狙った箇所までガイドするガイドRNA(sgRNA)とDNAを切断するCas9の複合体がCRISPER-Cas9です。
分子のハサミ、CRISPR-Cas9を流用させた技術はどんどん開発されており、今回はその1つ、塩基編集技術をご紹介します。
英語名はBase Editingで、一塩基編集、ベースエディティングとも呼ばれます。
ずばり、「切らないゲノム編集」です。
CRISPR-Cas9との大きな違いは、DNA鎖を切断しないこと。そのため、CRISPR-Cas9が抱える、狙った箇所以外も切断してしまうオフターゲット効果の問題は最小限に抑えられています。
この技術はとても興味深いもので、生物本来のDNA修復機構が働かないようにし、自然な変異をうまく利用することで、DNAを切断することなく編集しているのです。
目次
CRISPER-Cas9の仕組みをおさらい
塩基編集に見ていく前に、ちょっとおさらいをしておきます。
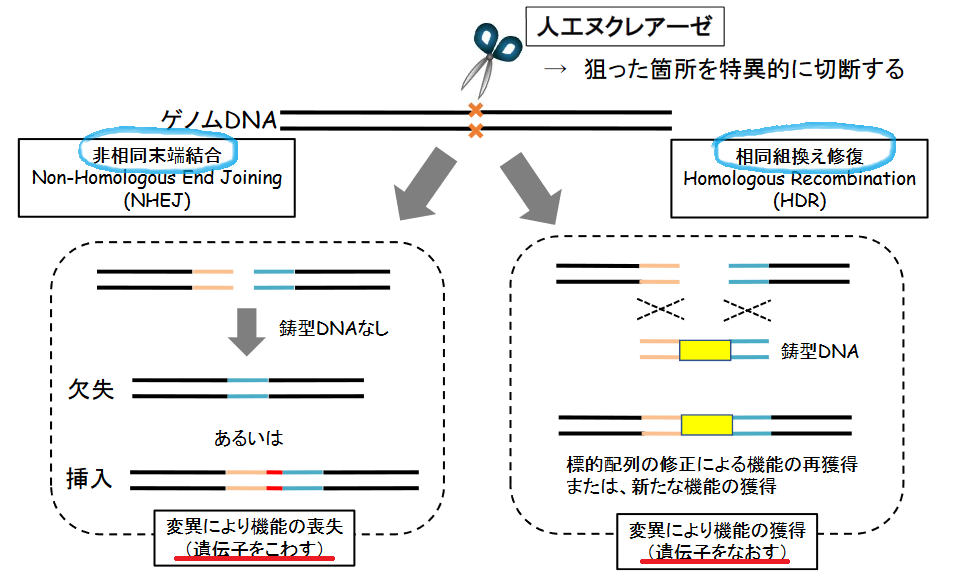
CRISPER-Cas9は、下記2つの二本鎖切断を修復する仕組みを利用します。
●非相同末端結合(NHEJ)
●相同組換え修復(HDR)
出典:ゲノム編集技術の現状(武田洋幸先生)https://www.mhlw.go.jp/content/10601000/000538826.pdf
簡単に書くと、
非相同末端結合は、切断された末端同士をつなぐものです。やっつけ仕事に近く、結合部位のヌクレオチドが追加されたり不足したりします。
相同組換えは、非相同末端結合で避けられない遺伝情報の損失を回避できる、より正確な修復です。
ただし、お手本となる鋳型が必要です。損傷されていないDNAが鋳型となり、損傷したDNA二本鎖が正確に修復されます。
この2つの仕組みをCRISPER-Cas9に利用すると、
●ハサミとなるCas9(人工ヌクレアーゼ)とガイドRNA(sgRNA)を導入(非相同末端結合)
➡標的遺伝子のある部分に欠損を起こさせ、変異を引き起こす
●ハサミとなるCas9(人工ヌクレアーゼ)とガイドRNA(sgRNA)+修復鋳型となるDNA断片を導入(相同組換え)
➡標的遺伝子の変異部分を修復したり、新たな機能を付与する
ざっくり言うと、
狙った箇所へ正確にたどり着いて切断するCRISPR-Cas9と、生物が本来もつDNA修復機構をうまくかけあわせて、ゲノムを編集します。
ただ、CRISPR-Cas9には、標的以外の類似配列を切断してしまうというオフターゲット効果という問題があります。
また、DNAの4つの塩基(A、T、C、G)のうち、1つだけほかの塩基に置換される点変異の修正では、効果が安定していませんでした。
そこでハーバード大学のDavid Liu氏によって開発されたのが、塩基編集(Base Editing)です。
Liu氏らの塩基編集技術で最初に開発されたのが、C–G塩基対をT–A塩基対へ変える方法です。
この塩基編集技術は、DNA二本鎖を切断することなく、官能基変換反応を利用して、塩基を変えているのが特徴です。
塩基編集(Base Editing)の仕組み
塩基編集には、大きく2つの変換パターンがあります。
●G-C ➡ T-A
●A-T ➡ G-C
今回は【G-C】➡【T-A】に変換するタイプをご紹介します。
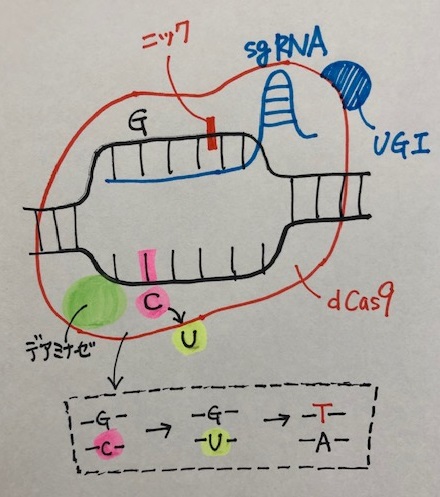
下のように、【C】を【U】に変えて、塩基を編集しています。どうやるのでしょうか?
https://www.the-scientist.com/modus-operandi/plant-genome-editors-get-a-new-tool-65368
塩基編集では、次の3点がポイントになります。
塩基編集のポイント
①Cas9(DNA切断酵素)を”切れないハサミ”にする
①官能基反応を利用
②本来のミスマッチ修復反応を阻害
おさらいですが、CRISPR-Cas9は、sgRNAとCas9の複合体です。
Liu氏らは、ハサミであるCas9を不活性にし、切れないハサミを開発しました。これを不活性型Cas9(dead Cas9、dCas9)といいます。
塩基編集ではdCas9を利用します。
このdCas9に、シトシン(C)をウラシル(U)に変換させるデアミナーゼという酵素を結合させています。
デアミナーゼとは
DNAの塩基を脱アミノ化する酵素
✔シトシンからウラシルへの変換反応
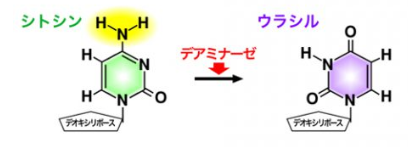
出典:NS遺伝子研究室(http://nsgene-lab.jp/technology/ge-applied-tehnology/)
dCas9に結合したデアミナーゼは標的配列に到着すると、塩基【C】を塩基【U】に変えます。
反応
デアミナーゼにより、G-C ➡ G-U となる

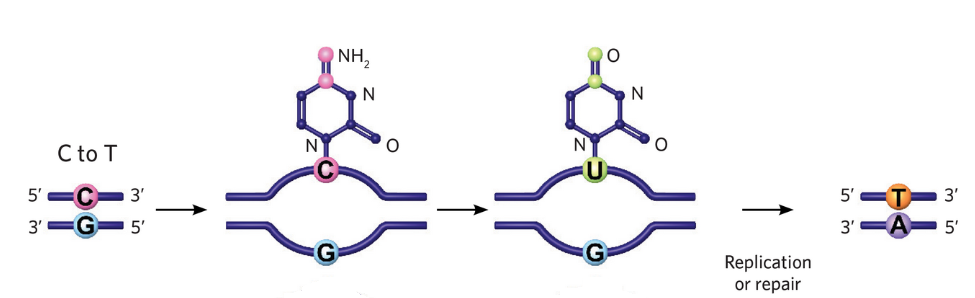
すると、DNAに一時的に【G-U】というペアが生じますが、【U】はDNAには存在しない塩基ですよね。
通常であれば、このようなミスマッチはウラシルDNAグリコシラーゼ(UNG)によって修復されます(ミスマッチ修復といいます)。
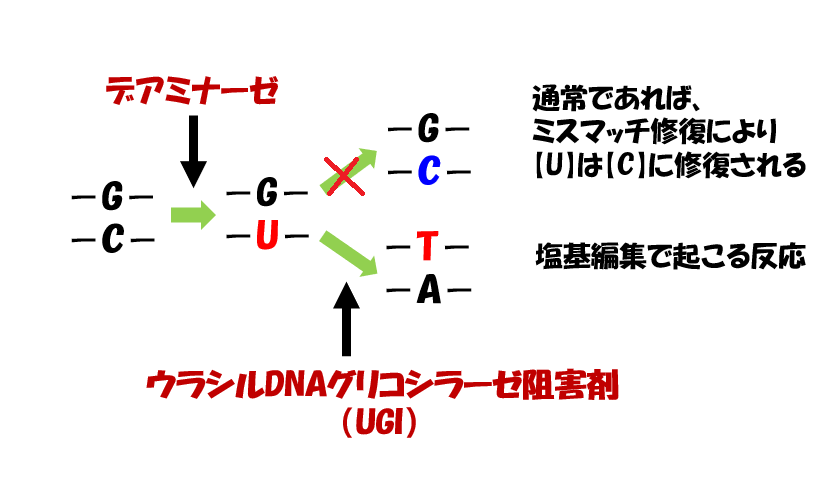
Liu氏らは、dCas9にウラシルDNAグリコシラーゼの阻害剤(UGI)を結合させました。
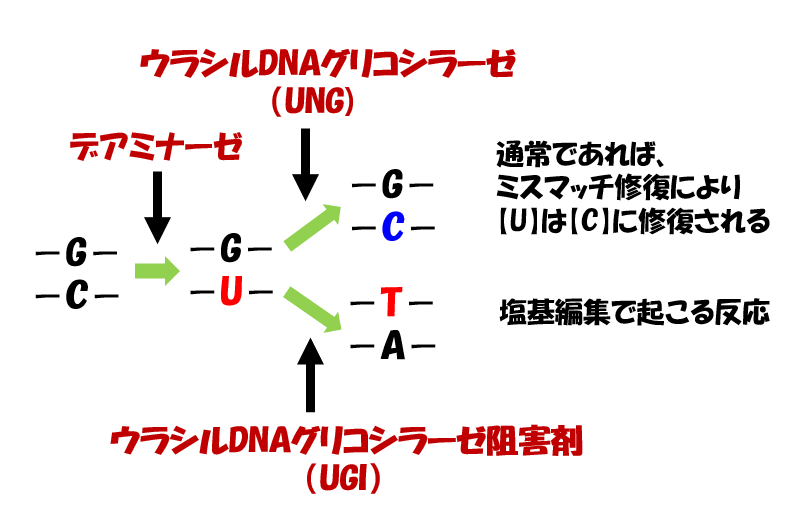
ウラシル【U】が【C】に修復されるのを防ぐ仕組みを導入したのです。
UGIを使うと、ウラシルDNAグリコシラーゼの働きが阻害され、【U】は【C】に戻れず、【U】のままDNAが複製されていきます(下図参照)。
【G-U】のペアのまま複製が進むと、2回目の複製で【T-A】が登場しますね。
このように、塩基編集では、生物がもつ本来のミスマッチ修復が起こらないようにして、塩基を変換させています。
塩基編集技術のもう1つの工夫
ゲノム編集効率を高めるために、Liu氏はニックを利用しています。
ニックとは、真核生物のミスマッチ修復で利用されるといわれる目印のことです。
真核生物では、2本鎖のうち、新しい鎖だけに現れるニックという切れ目を目印に、どちらの鎖の塩基を修復するべきか判断していると言われています。
出典:Chapter 5 DNA Replication, Repair, and Recombination (Slideshare)を引用、追記
※これはエッセンシャル細胞生物学の第3版に書かれていることですが、第4版では記載がありません。正確なメカニズムはわかっていないと考えられます。
Liu氏らは、脱アミノ化された塩基の反対の鎖(塩基が変換されていない鎖)にニックを入れています。
こうすると、脱アミノ化塩基の正常な修復が回避され、編集効率が高まるようです。

これは私の憶測ですが、編集していない鎖にニックを入れることで、修復機構を誤誘導し、人工的に変換した塩基が修復される可能性をさらに低くしているのではないかと思います。
塩基編集(Base Editing)のまとめ
【G-C→T-A】の場合
①Cas9(DNA切断酵素)を”切れないハサミ”(dCas9)にする
①官能基反応を利用して、シトシン(C)をウラシル(U)に変える
②本来のミスマッチ修復反応を阻害(dCas9にウラシルDNAグリコシラーゼの阻害剤(UGI)を結合させる)して、【T-A】に変える
世間ではゲノム編集食品に対する不安が強く、問題視されていますが、個人的にはゲノム編集技術に強い興味があります。
きっかけはこちらの2冊でした。
科学的に判断できるようになりたいので、引き続きゲノム編集について勉強していきます。
古いですが、こちらも買いました。
CRISPR-Cas9を流用させた技術はどんどん開発されているようですね。
今回は、その先駆者であるハーバード大学のDavid Liu氏の塩基編集をご紹介しました。
【参考】
ゲノム編集技術の現状 武田洋幸 第2回 ゲノム編集技術等を用いたヒト受精胚等の臨床利用のあり方に関する専門委員会